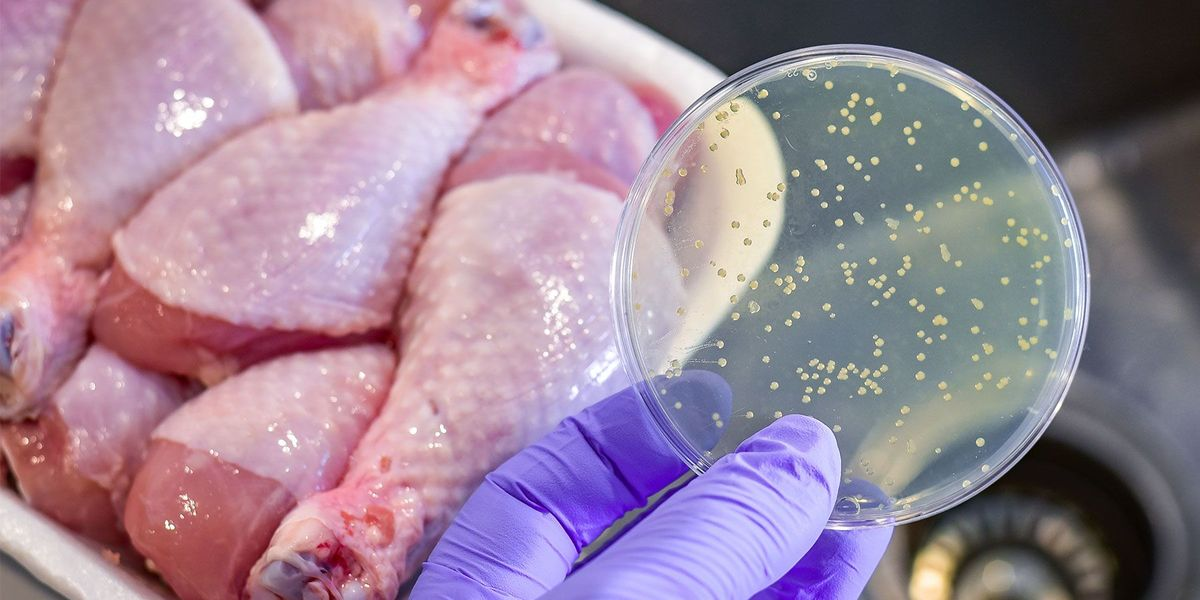
Daging unggas mentah merupakan salah satu penyebab utama keracunan Salmonella , yang menyerang ribuan orang di AS setiap tahunnya. Sebuah studi baru dari University of Illinois Urbana-Champaign menunjukkan bahwa beberapa produk dengan tingkat strain Salmonella yang sangat ganas yang tinggi bertanggung jawab atas sebagian besar penyakit yang berasal dari bagian daging ayam mentah. Para peneliti menyarankan agar upaya regulasi difokuskan pada pendeteksian dan pencegahan jenis kontaminasi berisiko tinggi tersebut.
“Selama 20 tahun terakhir, industri unggas telah berhasil menurunkan frekuensi Salmonella pada unggas. Namun, jumlah orang yang sakit akibat patogen ini tidak menurun. Departemen Pertanian AS sedang mempertimbangkan perubahan pada cara mereka mengatur Salmonella berdasarkan tingkat dan serotipe, dan penelitian kami mendukung upaya tersebut,” kata rekan penulis studi Matt Stasiewicz, seorang profesor madya di Departemen Ilmu Pangan dan Nutrisi Manusia (FSHN), bagian dari College of Agricultural, Consumer and Environmental Sciences (ACES) di Illinois.
Terdapat lebih dari 2.600 serotipe, atau subkelompok, bakteri Salmonella dan masing-masing berbeda dalam kemampuannya membuat orang sakit. Salmonella Kentucky merupakan salah satu serotipe yang paling umum pada ayam di AS, tetapi lebih kecil kemungkinannya menyebabkan penyakit pada manusia dibandingkan dengan tiga galur yang lebih ganas yang terkait dengan beberapa wabah salmonellosis.
Para peneliti ingin menilai risiko kesehatan masyarakat akibat kontaminasi Salmonella pada bagian ayam, dengan membandingkan dampak serotipe virulensi tinggi dan rendah pada tingkat yang berbeda.
Ia melakukan penelitian ini sebagai mahasiswa doktoral di FSHN, dan sekarang bekerja sebagai peneliti pascadoktoral di Layanan Penelitian Pertanian USDA.
Perhitungan dasar menghasilkan perkiraan sekitar dua kasus salmonellosis per 1 juta porsi ayam yang dikonsumsi. Dalam semua skenario, risiko terkonsentrasi pada beberapa produk dengan tingkat serotipe yang sangat virulen. Kurang dari 1% penyakit disebabkan oleh Salmonella Kentucky, sementara 69% hingga 83% penyakit disebabkan oleh produk dengan tingkat serotipe Enteritidis, Infantis, atau Typhimurium yang tinggi. Temuan ini konsisten dengan apa yang tampaknya mungkin menjadi perubahan peraturan yang diusulkan, kata para peneliti.
Langkah selanjutnya adalah mencari tahu cara untuk secara khusus menargetkan strain virulen tersebut. Kim dan Stasiewicz menyarankan pendekatan yang memungkinkan seperti menggunakan kontrol pemrosesan statistik untuk memantau Salmonella , prosedur uji-dan-tahan untuk sejumlah produk, atau memvaksinasi ayam terhadap serotipe dengan virulensi tinggi.
Namun, mereka menekankan bahwa penelitian mereka berfokus pada estimasi risiko, dan terserah pada industri perunggasan — yang paling tahu cara meningkatkan prosesnya — untuk menemukan strategi guna mengelolanya.
“Penelitian kami membantu menyelaraskan regulasi dengan kesehatan masyarakat, dan kemudian industri akan menemukan cara yang tepat untuk melakukannya,” kata Stasiewicz. “Temuan ini mendukung inisiatif USDA untuk mengalihkan regulasi ke peristiwa kontaminasi tingkat tinggi dan berisiko tinggi daripada frekuensi deteksi. Saya harap ini akan membantu konsumen memahami bahwa ini adalah strategi yang baik yang dirancang untuk melindungi kesehatan masyarakat. Orang awam mungkin berpikir bahwa regulasi baru ini membebaskan industri dari tanggung jawab, karena regulasi tersebut hanya menargetkan patogen tertentu dan memungkinkan ayam yang terkontaminasi masuk ke tahap produksi. Namun, masuk akal untuk berfokus pada jenis yang sebenarnya membuat orang sakit.”
Stasiewicz mengatakan Anda dapat menganggapnya sebagai proses tiga langkah: sains menunjukkan di mana risikonya, pemerintah menetapkan kebijakan regulasi, dan industri mencari cara untuk mengelola pengurangan risiko.
Para peneliti menekankan bahwa konsumen harus tetap mengikuti pedoman keamanan pangan saat menyiapkan unggas, seperti mencuci tangan, menghindari kontaminasi silang, dan memastikan daging dimasak dengan benar.